诺贝尔生理学或医学奖获得者Barbara McClintock在玉米中发现了第一个可移动的基因元件——转座子。转座子由一个编码转座酶的开放阅读框 (ORF)和两侧为转座酶结合位点的两个末端重复序列的序列组成。按照转座方式的不同,转座子可分为复制型和非复制型。其中,非复制型转座子研究最为广泛。转座酶是执行转座功能的酶,通常由转座子编码,识别转座子两末端的特异序列,能把转座子从相邻序列中脱离出来,再插入到新的DNA靶位点,无同源性要求。这么神奇的转座酶都有哪些作用呢?今天就带大家一探究竟。
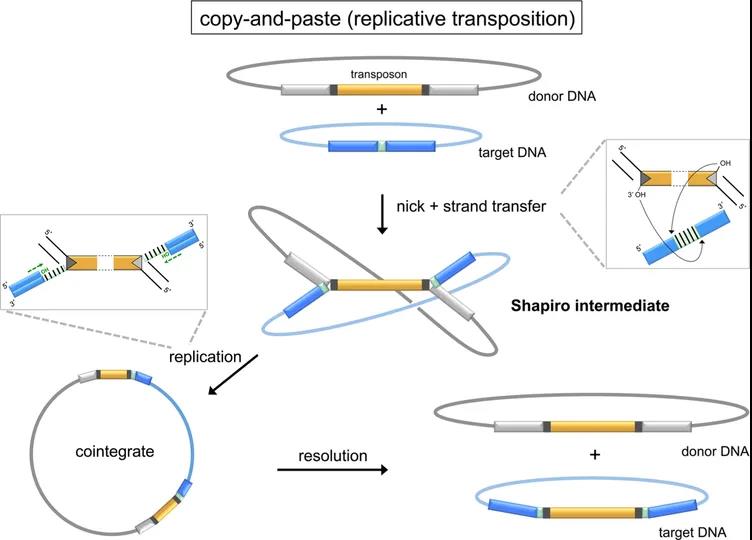
图1 复制型转座示意图
( Hickman A B, et al., Chemical Reviews, 2016)
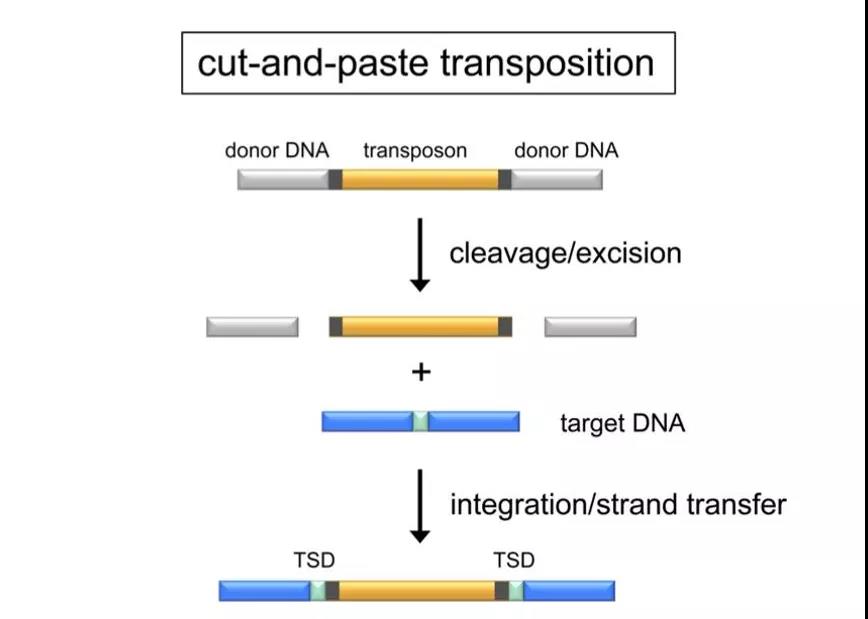
非复制型转座示意图
( Hickman A B, et al., Chemical Reviews, 2016)
转座子载体介导的稳转细胞系
转座子能够稳定地整合到基因组且在基因组中持久稳定表达。与具有穿越细胞膜并将其遗传物质传递到细胞核的内在能力的病毒载体相反,转座子是不能轻易穿过这些细胞屏障的移动 DNA 元件。因此,通常使用不同的非病毒策略递送到细胞中。与整合病毒载体相比,转座子具有较低的免疫原性,相对较大的载货容量以及致癌风险低等特点,这些特征使转座子成为有吸引力的基因传递系统之一。
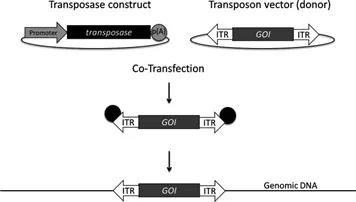
稳定的重组哺乳动物细胞在生物制剂的制药生物技术生产场景中越来越重要,例如单克隆抗体、生长和血液因子、细胞因子和亚单位疫苗。然而,经典的质粒 DNA 稳定转染建立重组细胞转染效率较低且单克隆细胞株的筛选耗时耗力。为了克服这些限制,在双组分 DNA 转座子衍生载体系统中,转座酶基因和 ITR 被分离到两个不同的质粒上。转座酶编码基因载体必须包含在所需宿主细胞中具有活性的合适启动子、编码转座酶的序列和位于3'的p(A)。供体载体包含感兴趣的基因序列(GOI), GOI两侧是ITR。在用两种质粒共转染靶细胞后,转座酶被表达,并从质粒骨架上切下由ITR构架的GOI片段,随后转座子载体稳定插入基因组中。
图3 双组分转座子载体系统
(Tschorn N , et al., Biotechnology Letters, 2020)
转座酶在基因组学研究中的应用
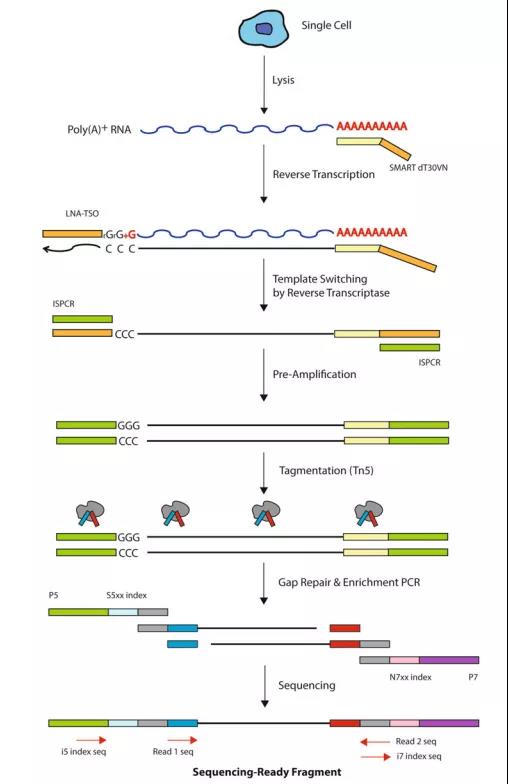
高通量测序(下一代测序技术(NGS))的发展和实验通量的不断提高,要求NGS的上游样品处理步骤尽可能简单,以提高整个NGS过程的效率。文库制备是一个重要的制约环节,NGS文库构建理论上为把双链DNA片段化为指定长度的序列,然后加上Read1和Read2、Index、P5和P7接头序列,可用于后续上机测序。研究人员发现,整个Tn5转座子序列不是转座必须的,只需转座子的末端核心序列,转座酶便能够将该部分序列插入基因组中,基于此原理,将测序接头序列加入末端核心序列中,转座酶切割双链DNA,然后把接头序列连接在DNA片段的两端,完成文库构建。传统的Illumina NEB建库方式需要经过DNA片段化、末端修复、接头连接、文库扩增、多次纯化分选等步骤,耗时较长,而转座酶构建NGS文库,可将DNA片段化、末端修复、接头连接等多步反应简化为1步反应,极大缩短建库时间,提高工作效率。基于转座酶建库原理,Smart-seq2可以实现单细胞全长转录组测序,能够揭示单个细胞的基因表达谱,反应细胞间异质性。
图4 Smart-seq2建库流程
(Simone, Picelli. Pubmed, 2019)
CRISPR相关转座酶系统
实现大片段DNA定向整合
定向DNA 整合被广泛用于基础研究和商业应用,因为它不仅可以保证外源DNA的安全,而且有助于在不受内部遗传变化干扰的情况下进行功能遗传研究。同源重组系统(HR)介导的基因插入方法目前被大多数人所推崇,其依赖于Cas核酸酶在靶点处发生DNA双链断裂(DSB),然后通过双交叉整合 ssDNA 或 dsDNA 供体到基因组中。但目前基于CRISPR/Cas系统的基因编辑仍然有不足之处,例如编辑效率低、整合片段大小有限、不能多位点同时整合等。
为了解决这些缺点,科学家们发现了两种新的CRISPR转座酶系统,新系统可在sgRNA的引导下无需Cas核酸酶活性即可实现大片段DNA在细菌基因组中的定向整合。张锋实验室使用来自蓝细菌Scytonema hofmanni的 CRISPR 相关转座酶 (CAST),它由 Tn7 -like转座酶亚基和 VK CRISPR 效应蛋白 (Cas12k) 组成。ShCAST 通过在原始间隔区下游 60–66 bp处单向插入DNA 片段来催化 RNA 引导的 DNA 转座,提供了一种不依赖于同源重组系统(HR)的定向插入 DNA 的策略。
图5 蓝细菌来源的CRISPR转座酶系统原理图
(Strecker, J. et al., Science,2019)
Samuel H. Sternberg实验室使用霍乱弧菌转座子Tn6677 可编码I-F CRISPR–Cas系统,该系统介导的DNA插入片段可在靶序列的正反向整合插入。接着Samuel H. Sternberg实验室在之前研究的基础上对I-F CRISPR–Cas系统进行优化,该系统整合效率为100%,可实现大片段DNA(高达10kb)的多位点定向整合。该系统在生物医学和工业相关细菌中均有特异性整合,为原核生物的大片段DNA定向整合提供了更多可能。
图6 霍乱弧菌来源的CRISPR转座酶系统原理图
(Vo P , et al., Nature Biotechnology, 2020)
参阅文献
1. Mario Di Matteo, Eyayu Belay, Marinee K Chuah & Thierry VandenDriessche. Recent developments in transposon-mediated gene therapy[J]. Expert Opinion on Biological Therapy, 2012, 12(7):841-858.
2. Su Bin Moon, Do Yon Kim Jeong-Heon Ko & Yong-Sam Kim.Recent advances in the CRISPR genome editing tool set[J]. Experimental & Molecular Medicine, 2019, 51(11):1-11.
3. SE Klompe,PLH Vo,TS Halpin-Healy,SH Sternberg. Transposon-encoded CRISPR–Cas systems direct RNA-guided DNA integration. Nature 571, 219–225 (2019).
4. Jonathan Strecker , Alim Ladha , Zachary Gardner , Jonathan L Schmid-Burgk , Kira S Makarova , Eugene V Koonin , Feng Zhang . RNA-guided DNA insertion with CRISPR-associated transposases. Science 365, 48–53 (2019).
5. Natalie Tschorn , Karen Berg , Jörn Stitz. Transposon vector-mediated stable gene transfer for the accelerated establishment of recombinant mammalian cell pools allowing for high-yield production of biologics[J]. Biotechnology Letters, 2020, 42(Suppl 1).
6. Alison B Hickman , Fred Dyda. DNA Transposition at Work[J]. Chemical Reviews, 2016.
7. Grunenwald, Haiying Li,Caruccio, Nicholas,Jendrisak, Jerome,Dahl, Gary. Transposon end compositions and methods for modifying nucleic acids:, EP2508529[P]. 2013.
8. Simone, Picelli. Full-Length Single-Cell RNA Sequencing with Smart-seq2.[M]. Pubmed, 2019.
该文章来自于网络转载,版权归原作者所有。如有涉及侵权请告知,我们将尽快删除相关内容。